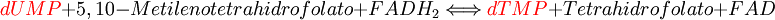|
Los desoxirribonucleótidos son los monómeros que constituyen el ADN. Y poseen la misma estructura que los nucleótidos :
La gran diferencia entre un ribonucleótido y un desoxirribonucleótido se encuentra en la molécula de azúcar (ribosa y desoxirribosa, respectivamente). Esta imagen nos muestra la diferencia entre las moleculas de azucar. A la izquierda se encuentra la ribosa, y señalado con una flecha, se encuentra el grupo funcional hidroxilo (OH), que no se encuentra en la desoxirribosa (derecha). Es justamente por este motivo que se le llama Desoxi-ribosa, ya que no posee ese grupo hidroxilo, y por ende, a los nucleótidos que incorporan esta azúcar a su estructura se les denomina desoxirribonucleótidos. Técnicamente, el carbono indicado con la flecha, es llamado el Carbono 2' o 2'-C, debido a convenciones químicas de prioridad atómica en moléculas cíclicas (o sea, anillos). Cuatro son las base nitrogenadas presentes en los desoxirribonucleótidos: adenina, timina, guanina y citosina. El uracilo forma parte de los ribonucleótidos y la timina esta presente en los desoxirribonucleótidos, y en muy raras ocasiones se presenta el caso contrario.
Conocimientos adicionales recomendados
NomenclaturaPara diferenciar los ribonucleotidos de los desoxirribonucleotidos, existe la siguiente nomenclatura que puede aparecer en documentos más especializados.
EstabilidadA pesar que tanto el ADN como el ARN son moléculas estables, el ADN lo es mucho más. Esto ocurre por la utilización de desoxirribonucleótidos en vez de ribonucleótidos en su síntesis, aunque no exclusivamente por esa razón.[1] El ARN, debido a que usa ribosa y posee el grupo hidroxilo, es susceptible a hidrólisis catalizada por bases. Si el grupo 2'-OH fuese sacado de la molecula, la tasa de hidrólisis del ARN baja unas 100 veces en condiciones neutras.[2] De esta manera, es posible pensar que la remoción del grupo hidroxilo en la ribosa fue un agente importante en permitir la complejidad de organismos que vemos hoy en día. Conversion de ribonucleótidos a desoxirribonucleótidosVéase también: Ribonucleótido reductasa
Los desoxirribonucleótidos provienen de los ribonucleótidos, al reducir el Carbono 2' del anillo de ribosa, mediante la enzima ribonucleótido reductasa.[3] Esta enzima utiliza nucleótidos di-fosfatados (NDP) para catalizar su reacción:[4]
![NDP \xrightarrow[reductasa]{Ribonucleotido} dNDP + H_2O](images/math/4/d/8/4d83871bfc2a892cda2b0493a2769b5d.png) La ribonucleótido reductasa al catalizar esta reacción, pasa a un estado oxidado inactivo. Sin embargo, ésta puede recobrar su poder reductor mediante dos vías: La glutarredoxina, que actúa a través del glutatión o la tiorredoxina, que actúa mediante transferencia de hidrógenos de NADPH2 a través de un grupo prostético FAD. Timina: su uso y síntesisVéase también: Reparación del ADN y Timidilato sintetasa
El uso de timina en el ADN, en vez de utilizar uracilo, puede deberse a una presión selectiva que ayude a mantener la integridad de la secuencia genética, puesto que el grupo metilo en la timina, facilita la reparación de ADN.[2] Existen dos formas para sintetizar timina: Utilizando como precursor CDP (citidina difosfato), o bien UDP (uridina difosfato). En ambos casos, la transformación final a dTMP es catalizado por la enzima timidilato sintetasa,[5] cuya reacción es la siguiente:[6]
Vía del CDPSolo posee un paso más que la vía del UDP, ya que el precursor directo de la timina es el uracilo. La vía es la siguiente: 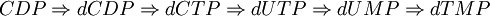 Vía del UDPLa única diferencia es que no posee el paso de dCTP a dUTP. La via es la siguiente: 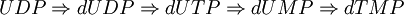
Referencias
Veáse tambiénCategoría: Nucleótidos |
||||||||||||||||
| Este articulo se basa en el articulo Desoxirribonucleótido publicado en la enciclopedia libre de Wikipedia. El contenido está disponible bajo los términos de la Licencia de GNU Free Documentation License. Véase también en Wikipedia para obtener una lista de autores. |


Enciclopedia