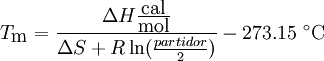|
Producto destacadoEn la mayoría de replicaciones del ADN, el principal partidor para la síntesis de ADN es una cadena corta de ARN. Este ARN lo produce una ARN polimerasa, y luego una ADN polimerasa lo elimina y lo sustituye con ADN. Muchas técnicas de laboratorio en biología molecular que utilizan ADN polimerasas necesitan partidores; técnicas como la secuenciación de ADN y la reacción en cadena de la polimerasa (PCR). Los partidores usados para estas técnicas usualmente son moléculas de ADN cortas y sintetizadas de forma química, de aproximadamente veinte bases de longitud. La construcción en si de estos partidores empieza con nucleósidos de 3'-hidroxil (phosphoramidite) adheridos a un material llamado vidrio de poros controlados (CPG por sus siglas en inglés). El 5'-hidroxil de los nucleósidos es dimetoxitritilo (DMT) cubierto, lo que previene la construcción de una cadena de nucleótidos. Para añadir un nucleótido se remueve químicamente el DMT y se añade el nucleótido. El 5'-hidroxilo del nuevo nucleótido queda bloqueado por el DMT, lo que evita la adición de más de un nucleótido a cada cadena. Después de eso se repite el ciclo para cada nucleótido en el partidor. Esta es una descripción simplificada - el proceso en realidad es bastante complicado. Por esta razón la mayoría de laboratorios no construyen los partidores sino que los compran a empresas especializadas. La secuenciación de ADN se usa para determinar los nucleótidos en una cadena de ADN. Un método de secuenciación llamado secuenciación didesoxi, conocido también como método de terminación de cadenas o método Sanger, usa un partidor como marcador de inicio para la reacción de cadena. En la reacción en cadena de la polimerasa se usan partidores para determinar el fragmento de ADN que será amplificado en el proceso de PCR. La longitud de los partidores no suele ser mayor de 50 nucleótidos (la longitud se mide en pares de bases, ya que el ADN usualmente es de doble filamento. La longitud del ADN de un solo filamento se mide en bases o nucleótidos), y son iguales al inicio y al final del fragmento de ADN que va a ser amplificado. Se adhieren a la plantilla del ADN en estos puntos de inicio y fin a los cuales se une la ADN polimerasa y la síntesis de la nueva cadena empieza. Diseño de partidoresLa elección de la longitud de los partidores y de la temperatura a la que se derriten (Tm) depende de varias consideraciones. La temperatura de derretimiento de un partidor se define como la temperatura a la cual el 50% de esa misma especie de molécula de ADN forma una doble hélice estable y el otro 50% se separa en moléculas de un solo filamento. La temperatura de derretimiento requerida aumenta con la longitud del partidor. Los partidores que son demasiado cortos se anexarían en diversas posiciones en una larga plantilla de ADN, lo cual llevaría a copias no específicas. Por otro lado, la longitud del partidor está limitada por la temperatura requerida para derretirlo. Las temperaturas de derretimiento muy altas, es decir por encima de los 80°C pueden causar problemas porque la ADN polimerasa es menos activa en esas temperaturas. La longitud óptima de un partidor generalmente está entre 20 y 30 nucleótidos con una temperatura de derretimiento de entre 55 y 65 °C. Hay muchas formas de calcular la temperatura de derretimiento de los partidores (A, G, C y T son el número de nucleótidos en el partidor, respectivamente. [Na+] es la concentración de Na+ en el tubo de PCR).
El paquete de software libre primou puede calcular la temperatura de anexión de acuerdo con el método de apilamiento de bases. Un partidor no debería anexarse fácilmente a sí mismo u otros de su tipo, con lo cual se producirían bucles o pinzas. Esto podría entorpecer la anexión con la plantilla de ADN. Sin embargo, normalmente las pinzas son inevitables. Algunas veces se usan partidores degenerados. Estos en realidad son mezclas de partidores similares pero no idénticos. Pueden ser convenientes si se va a amplificar el mismo gen de organismos, puesto que los genes mismos pueden ser similares pero no idénticos. El otro uso para los partidores degenerados es cuando el diseño de partidores se basa en la secuencia de proteínas. Como muchos codones diferentes pueden codificar un aminoácido suele ser difícil deducir qué codón se usa en un caso particular. Por ello la secuencia de partidores que corresponde al aminoácido isoleucina podría ser "ATH", por A de adenina, T de timina y H de adenina, timina o citocina, según el código genético de cada codón. El uso de partidores degenerados puede reducir ampliamente la especificidad de la amplificación por PCR. El problema puede resolverse usando PCR touchdown. Vínculos extenos
Categoría: Bioquímica |
| Este articulo se basa en el articulo Partidor publicado en la enciclopedia libre de Wikipedia. El contenido está disponible bajo los términos de la Licencia de GNU Free Documentation License. Véase también en Wikipedia para obtener una lista de autores. |





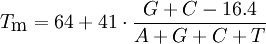
![T_\mbox{m}=100.5+41 \cdot \frac{C+G}{A+C+G+T}-\frac{820}{A+C+G+T} \cdot 16.6 \cdot \log_{10}([\mbox{Na}^+])](images/math/d/b/a/dba42ed0e79ae00dbbb8bc46621d15cc.png)